目的
データ分析の仕事をする中で最も扱う機会が多いのが
時系列データだと思います。その中で欠損値を扱ったり、
統計を取ったり、特徴量を作り出したりするのですが、
毎回やり方を忘れてググっているので、上記の書籍を読んで
こういった前処理の方法をいつでも確認できるように
メモしておこうと思います。
目次
- 目的
- 目次
- 日時のデータをdatetime型に変換する
- 最初の日時からの経過時間を計算する
- 各データの統計量を計算する
- 欠損値の確認と補完
- 経過時間の単位を変換する
- データフレーム結合する
- 基準日時からの経過時間を計算する
- 重複した行を削除する
- 特定のデータ列をインデックスにする
- 部分的時系列を抽出して統計量を計算する
- データフレームの各列をリストにして結合する
- 不均衡データから教師データを作成する
- オートエンコーダで教師データを作成する
日時のデータをdatetime型に変換する
解析したいデータが時系列で記録されたCSVファイルがあり、
それを読み込んでデータフレームにしたとします。
そのデータフレームにある各データの型をこのコードで確認すると、
print(data_df.dtypes)
各データの型がこのように出力されます。

日時のデータにあたるdateの型はobject型となっているので、
このままでは解析しにくいです。そこで、こちらのコードで
datetime型へと変換することで扱いやすくなるようにします。
data_df["date"] = pd.to_datetime(data_df["date"], format="%Y-%m-%d %H:%M:%S") print(data_df["date"].dtypes) print(type(data_df["date"][0]))
これにより、dateの列はdatetime64型、date列の要素はTimestamp型と
なることが確認できます。
![]()
最初の日時からの経過時間を計算する
まずは各行の時間の差分をこのようなコードで計算します。
data_df["dif_min"] = data_df["date"].diff().dt.total_seconds() / 60 data_df["dif_min"] = data_df["dif_min"].fillna(0) print(data_df["dif_min"].head())
これにより、先頭行の時間から順に10分間隔でデータが記録
されていることが分かります。

そして、計算した行間の時間の差分を利用して、このコードに
より累積和を求めることで経過時間を得ることができます。
data_df["cum_min"] = data_df["dif_min"].cumsum() print(data_df[["date", "cum_min"]].head())
日時と経過時間

各データの統計量を計算する
データフレームに記録されている各データについて、
下記の統計量を計算することができます。
- count: 件数
- mean: 平均値
- std: 標準偏差
- min: 最小値
- 25%: 第一四分位数
- 50%: 第二四分位数、中央値
- 75%: 第三四分位数
- max: 最大値
計算するためのコード
data_df.describe()
計算結果

欠損値の確認と補完
大規模な時系列データを見ると、データによって記録される周期が
異なり、データフレームにすると欠損がある行が見つかる場合が
あります。まずは元のデータに欠損がないかをこちらのコードで
確認できます。
print(data_df.isnull().sum(axis=1).sort_values(ascending=False)) print(data_df.isnull().sum(axis=0))
1行目のコードでは各行における欠損値の個数を確認しています。

2行目では各列における欠損値の個数を確認しています。

ここまでのコードで欠損値の存在を確認できたので、今度は補完する
処理を行います。時系列データはその並びに意味があるので、単純に
0や定数、平均値などで補完してはいけません。なので、前の行の値や
前後の値に基づいて補完するのが適しています。
前の行の値を代入することで補完する場合は下記のコードでできます。
data_df[initial_index:end_index].fillna(method="ffill")
また、こちらのコードのようにすれば、前後の行の値の平均値で
補完することができます。
data_df = data_df.interpolate()
経過時間の単位を変換する
例えば、もともとのデータが10分単位で記録されたものだとして、
それを1時間単位で集計したい場合、こちらのコードでまずは、
1時間単位で連番を付与した列を作ります。
data_df["cum_hour"] = (data_df["cum_min"] / 60).round(2).astype(int) print(data_df[["date", "cum_min", "cum_hour"]].head(10))
実行するとこのように、最初は0で時間が1時間経過してから1となる
列が作成されます。

そして、上記のコードで作成される"cum_hour"をキーとすることで、
1時間毎にデータを集計した平均値と標準偏差を計算できます。
print(data_df.groupby("cum_hour").mean()) print(data_df.groupby("cum_hour").std())
1時間毎の平均値

1時間毎の標準偏差

データフレーム結合する
pandasのmerge()を使うことで、2つの異なるデータフレームを
結合することができます。
data_merged = pd.merge(data_df_1, data_df_2)
また、ここでインデックスをキーにして結合する場合は、
left_indexとright_indexというオプションをTrueにします。
data_merged = pd.merge(data_df_1, data_df_2, left_index=True, right_index=True)
基準日時からの経過時間を計算する
1行目のコードで、日時が記録されてる列を指定のフォーマットで
datetime型のデータに変換します。次に2行目のように基準とする
日時を決め、3行目で各行の日時との差分を計算します。この時、
基準とする日時もdatetime型に変換しないと差分が計算できない
ので注意しないといけません。そして最後に4行目で、計算した
経過日時を経過秒→分と変換することで経過時間が求まります。
event_df["date"] = pd.to_datetime(event_df["date"], format="%Y-%m-%d %H:%M:%S") base_time = "2016-01-11 17:00:00" event_df["dif_min"] = event_df["date"] - dt.datetime.strptime(base_time, "%Y-%m-%d %H:%M:%S") event_df["dif_min"] = event_df["dif_min"].dt.total_seconds() / 60
重複した行を削除する
data_df = data_df[~data_df.duplicated()]
特定のデータ列をインデックスにする
data_df = data_df.set_index(["data"])
部分的時系列を抽出して統計量を計算する
例えば10分毎にデータが記録されたデータフレームがあるとし、
1行ずつずらしながら一定期間のデータを抽出して統計量を
計算したい場合はこのようなコードで実現できます。
data_df = data_df.rolling(6).mean()
rollingを使ってデータを1行ずつスライドさせていく窓を作成し、
それにより抽出されたデータの平均値を計算しています。
rollingへの引数には窓によって抽出する行数を指定します。
このとき、先頭行から5行目までは欠損値となるため、
このようにしてデータフレームから除外しておきます。
data_df = data_df.dropna()
データフレームの各列をリストにして結合する
data_list = (np.array(data_df["data1"]).tolist() + np.array(data_df["data2"]).tolist() + np.array(data_df["data3"]).tolist())
不均衡データから教師データを作成する
「いつ異常が発生したか」をデータから学習したい場合、
異常の発生はそう多くはないため、正常時のデータと比べて
異常時のデータはとても少ないケースがほとんどです。
そういうときは、これから述べるやり方で異常発生の兆候を
数値化して教師データとすることができます。
データの読み込みと可視化
まずは全ての時系列データが記録されたCSVファイルを読み込み、
その中から日付に関するデータと異常の兆候とするデータのみを
抽出してデータフレームにします。このとき、日付データは
datetime型に変換しておきます。
data_df = pd.read_csv("data.csv", sep=',')[["date", "data1"]] data_df["date"] = pd.to_datetime(data_df["date"], format="%Y-%m-%d %H:%M:%S")
読み込んだデータを試しに可視化してみます。
このとき、日付をx軸にしたい場合はこのコードのように
ラベルを回転させることで見やすくできます。
plt.plot(data_df["date"], data_df["data1"]) plt.xlabel("date") plt.xticks(rotation=30) plt.ylabel("data1") plt.show()

K近傍法で教師データを作成する
ここでは、K近傍法(K Nearest Neighbor)により教師データを
作成する手順について説明します。ここで言う教師データとは、
各データに対して正常か異常かをラベリングしたデータセット
のことを指します。
K近傍法とはクラス判別手法の一つであり、得られた未知のデータ
から近い順にデータをK個取得し、その多数決でどのクラスに
属するかを推定するというものです。
まずは元のデータを、訓練用データとテスト用データに分けます。
分け方にもいろいろありますが、ここではある日付を基準にして
分けてみます。
train = data_df[data_df["date"] < "2016-04-11 17:00:00"] test = data_df[data_df["date"] >= "2016-04-11 17:00:00"]
また、データの値が0から1の範囲になるように正規化しておきます。
from sklearn.preprocessing import MinMaxScaler mc = MinMaxScaler() train = mc.fit_transform(train[["data1"]]) test = mc.fit_transform(test[["data1"]])
正規化についての詳しい記事
atmarkit.itmedia.co.jp
正規化ができたら、次はスライド窓により部分的時系列を作成します。
ここでのスライド窓の幅は、1日分のデータをカバーするサイズと
し、それを1データずつずらしながら部分的時系列を作っていくという
処理をこちらのコードで行います。
width = 144 train, test = train.flatten(), test.flatten() train_vec, test_vec = [], [] for i in range(len(train) - width): train_vec.append(train[i:i + width]) print(pd.DataFrame(train_vec).shape) print(pd.DataFrame(train_vec).head()) for i in range(len(test) - width): test_vec.append(test[i:i + width]) print(pd.DataFrame(test_vec).shape) print(pd.DataFrame(test_vec).head())
1行目のwidthがスライド窓の幅です。ここで2行目により、訓練データと
テストデータはflatten()により1次元配列にしておきます。そして、
そこから1データずつずらしながらスライド窓の幅分のデータを抽出し、
配列に詰めていくという処理を3行目以降で行っています。
ここまでの処理によって作成される訓練データの部分的時系列は
こちらのようになります。

スライド窓により抽出された144個のデータが横並びになり、それが
1データずつずらしながら抽出した回数分だけ縦に並びます。同様に
テストデータに対しても実行するとこのようになります。

異常検出モデルを作成する
ここまでの前処理により訓練データの部分的時系列を作成できました。
続いて、このデータを用いてK近傍法による異常検出モデルの作成を
こちらのコードで行います。このコードでは、K=1として最も近いデータとの距離を測るモデルを作成しています。
train_vec = np.array(train_vec)
model = NearestNeighbors(n_neighbors=1)
model.fit(train_vec)
そして、このモデルをテストデータに適用し、各テストデータと
最も近い訓練データとの距離を求めます。実行するコードはこちらの
ようになります。
test_vec = np.array(test_vec) dist, _ = model.kneighbors(test_vec) dist = dist / np.max(dist) plt.plot(dist) plt.show()
2行目のコードで計算された距離が出力され、それを0から1に
収まるように正規化します。そして、それをプロットすると
このようになります。この距離の傾向に基づいて閾値を設定し
それを超えるなら異常、でなければ正常としてラベリングすれば、
正常か異常かを学習できる教師データセットを作成できます。

オートエンコーダで教師データを作成する
今回のような異常検出に利用されるもう一つの手法として、
オートエンコーダがあります。これは教師なし学習のアルゴリズム
であるため、正常時のデータのみで学習を行えます。
オートエンコーダの解説と活用事例
www.tryeting.jp
オートエンコーダの仕組み
オートエンコーダはニューラルネットワークにおける
ネットワークの形の一種です。入力層、隠れ層、出力層という
三つの層に分かれます。
特徴量の元になる説明変数が入力層に渡され、そこから隠れ層に
向けてデータの次元を圧縮し、最後に隠れ層から出力層に向けて
復元します。これにより、不要なデータをそぎ落として、意味の
あるデータだけを残して特徴量として扱えるようになります。
異常検出モデルを学習する
まずは、深層学習用パッケージであるKerasを用いて、このような
オートエンコーダ・ネットワークを作成するコードを実装します。
from keras.models import Sequential from keras.layers import Dense model = Sequential() model.add(Dense(128, activation="relu", input_shape=(144,))) model.add(Dense(64, activation="relu")) model.add(Dense(32, activation="relu")) model.add(Dense(64, activation="relu")) model.add(Dense(128, activation="relu")) model.add(Dense(144, activation="sigmoid")) model.summary()
4行目でSequencialモデルを作るためのインスタンスを
生成します。このとき、K近傍法でモデルを作成したときの
スライド窓の幅と同様に、144を特徴量の数としてinput_shapeに
セットします。
Kerasにおけるモデル作成方法について
qiita.com
続いて5~9行目で、Denseによる隠れ層の作成を行います。
各行では、層のノード数と活性化関数をパラメータとして指定
しています。
活性化関数について詳しく解説された記事
zenn.dev
そして10行目で、出力層を作成しています。
ここでのノード数は入力層と同様に144個とし、
活性化関数はシグモイド関数を指定しています。
作成したネットワークの内容は、summary()により確認できます。

今度は上記で作成したモデルの学習をこちらのコードで行います。
1行目では学習条件として、誤差関数と最適化手法を指定しています。
2~3行目で学習を行いますが、このときに入力する各引数は左から
順に、説明変数、目的変数、ミニバッチ数、学習進捗の表示形式、
エポック数、検証データに使用する比率を指定します。
model.compile(loss="mse", optimizer="adam") hist = model.fit(train_vec, train_vec, batch_size=128, verbose=1, epochs=20, validation_split=0.2)
実行するとこのように学習は行われます。

学習が終了したら、こちらのコードによりエポック数ごとの
誤差関数の収束具合を可視化することができます。
plt.plot(hist.history["loss"], label="loss") plt.plot(hist.history["val_loss"], label="val_loss") plt.ylabel("loss") plt.xlabel("epoch") plt.legend() plt.show()
誤差を可視化するとこのように、エポックを重ねるごとに
収束しているのが確認できます。

学習したモデルにテストデータを適用する
ここまででモデルを学習することができたので、
今度はこちらのコードにより、テストデータに
適用した場合の出力を検証してみます。
pred = model.predict(test_vec) plt.plot(test_vec[:, 0], label="test") plt.plot(pred[:, 0], label="pred") plt.legend() plt.show()
このようにテストデータの元の値とモデルによる出力値を
可視化してみると、元の値と同じような傾向を出力値も
示していることが分かります。
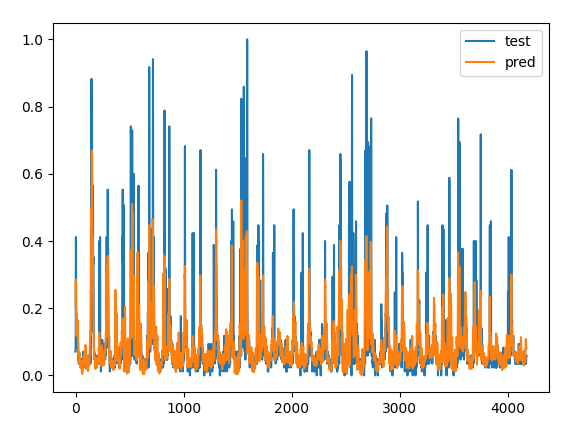
最後に、テストデータの元の値と、モデルによる出力値との
差分を、このコードで計算し可視化してみます。このとき、
差分は正負の値をとるため、二乗することで絶対値の大小で
比較できるようにします。また、0から1の範囲に収まるように
正規化もしておきます。
dist = test_vec[:, 0] - pred[:, 0] dist = pow(dist, 2) dist = dist / np.max(dist) plt.plot(dist) plt.show()
これにより計算される差分を可視化するとこのようになり、
この差分を異常スコアとして閾値処理することで、閾値より
大きければ異常、小さければ正常というラベリングが可能と
なります。

